Una de las cuestiones que más controversia suele causar en nuestra vida íntima es la de si el tamaño realmente importa o no (que por cierto, un tamaño excesivo puede resultar hasta contraproducente). Pero hoy no estamos aquí para hablar de diversión en la cama, sino de genomas y su complejidad (vaya, qué decepción).
Los que me siguieseis con anterioridad en la cuenta de Instagram (@elbiolocomolecular) me habréis visto hablar un montón de veces sobre genes y genomas, pero obviando un poco el significado de estos términos y asumiendo que la mayoría de lectores/as los entenderíais, pero hoy voy a ahondar un poco más en estos conceptos y voy a hablaros sobre si el tamaño del material genético determina o no la complejidad de los seres vivos.
Definiciones básicas: gen y genoma
En primer lugar, vamos a definir los conceptos clave de este post: gen y genoma.
La definición de gen ha ido evolucionando a lo largo del tiempo, y además puede variar entre autores/as, pero básicamente, un gen se puede definir como aquella secuencia de nucleótidos que contiene la información necesaria para la síntesis de un RNA o proteína funcional (esto no es exactamente así, pero definiremos de esta manera el gen para que sea fácil de entender) (1). Algunos autores también consideran que se debe incluir dentro del concepto de gen aquellas secuencias reguladoras que influyen en la expresión génica (como los promotores, secuencias que forman bucles o loops, operadores o enhancers) (2).
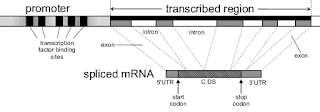
Estructura de un gen (Fuente). Vemos la región promotora corriente arriba del inicio de transcripción, y dentro de la región transcrita tenemos las secuencias no traducidas de los extremos 5' y 3' (5' & 3'-UTR), los exones (que formarán parte de la secuencia codificante (CDS) del mRNA maduro) y los intrones (que serán eliminados posteriormente).
Cuando hablamos del genoma nos referimos a todo el material genético que poseen las células de un organismo concreto, es decir, toda la secuencia de DNA que almacena la información que va a determinar las características del individuo y va a permitir que lleve a cabo las funciones necesarias para la vida (3). Dentro del genoma encontramos, obviamente, todo el conjunto de genes que van a permitir a las células sintetizar las proteínas necesarias para realizar sus funciones, pero como ya indicamos antes, no solo existen genes dentro del genoma, sino que también hay una enorme cantidad (alrededor del 99% del genoma humano (4)) de regiones no codificantes (que no dan lugar a proteína). A todo este DNA no codificante se le llamaba (erróneamente) "DNA basura", pero se ha visto que esas regiones no codificantes pueden desempeñar, por ejemplo, funciones reguladoras, como los ya mencionados promotores o los enhancers. Además, también podemos encontrar pseudogenes (secuencias muy similares a los genes activos, pero que han perdido su funcionalidad debido a mutaciones, inserciones, deleciones...) (5), intrones (regiones situadas dentro de un gen funcional que inicialmente se transcriben, pero que son eliminadas durante la formación de los mRNA maduros), secuencias que se transcriben dando lugar a RNAs reguladores (como los microRNAs o los lncRNAs) o transposones (fragmentos de DNA capaces de "saltar" de una región a otra del genoma, cuyo origen es principalmente vírico y de los que ya hablé en un post anterior en mi cuenta de Instagram).
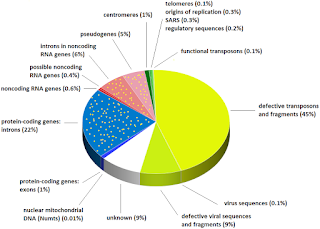
El genoma humano está constituido únicamente en un 1% de secuencias que codifican proteínas, y alrededor de un 9% del DNA total no se sabe siquiera qué función desempeña (Fuente).
De hecho, todos esos transposones (como el retrotransposón LINE-1) constituyen hasta casi la mitad de todo nuestro genoma -una locura, la verdad- y en ocasiones pueden ser los responsables de la aparición de tumores (6), ya que si deciden "saltar" a otro lado de nuestro genoma e insertarse dentro de genes (o en sus secuencias reguladoras) que codifican proteínas implicadas en el control del ciclo celular o la expresión génica, pueden provocar la aparición de fenotipos tumorales y que las células afectadas por estas inserciones se dividan sin control, dando lugar a diversos tipos de cáncer.
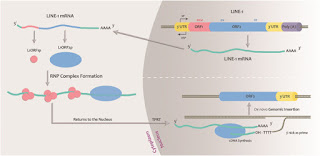
Esta es la forma en la que nuestro colega LINE-1 hace una copia de sí mismo, y se mete en la región del genoma que más rabia le dé en ese momento (7).
El genoma y las regiones no codificantes
Sin embargo, existen otros organismos que poseen una cantidad de DNA no codificante mucho menor, como es el caso de la planta carnívora Utricularia gibba, que tan solo posee un 3% de DNA no codificante (8). Si nos vamos a organismos procariotas (bacterias) vemos que la proporción de DNA no codificante es bastante menor que en eucariotas (sobre todo en comparación a los humanos), ya que los procariotas presentan una evolución más acelerada al tener una alta tasa de reproducción (9). Entonces, podemos observar cómo la cantidad de DNA no codificante determina en gran medida cuál va a ser el tamaño final del genoma (es decir, el número de pares de bases (bp) totales que lo constituyen), especialmente en organismos eucariotas, por lo que puede darse un fenómeno que se conoce como paradoja del valor C.
Aquí tenemos a Utricularia gibba, que no se anda con pijadas y pasa olímpicamente de tener demasiado DNA no codificante en su genoma (le gusta ir al grano, solo quiere genes codificantes y poco más, qué crack).
La paradoja del valor C hace referencia al hecho de que el tamaño del genoma puede no corresponderse a la aparente complejidad de un organismo. Vamos a traducir esto: en primer lugar, hemos de saber qué es el valor C. El valor C hace referencia a la cantidad de DNA (en picogramos) contenido en un núcleo haploide, por lo que es una medida del tamaño del genoma (haploide) de un organismo (10).
Lo primero que uno piensa es que, a mayor complejidad de un organismo, mayor tamaño tendrá su genoma, pero como ya hemos visto que en eucariotas la proporción de DNA no codificante, además de ser bastante considerable, varía mucho entre especies, no podemos aseverar que un organismo "sencillo" o menos desarrollado que otros posea un genoma de menor tamaño, ya que puede darse el caso de que posea una gran cantidad de regiones no codificantes que ampliarían en gran medida su valor C, y por tanto, el tamaño de su genoma en pb totales.
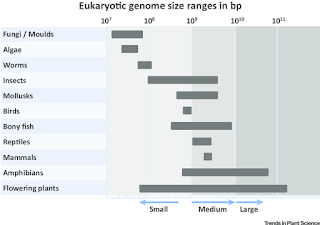
Como vemos en esta tabla, hay organismos (como las plantas con flores o angiospermas) que a pesar de ser aparentemente más sencillos que, por ejemplo, los mamíferos, poseen un genoma de mayor tamaño (Fuente).
Es más, aunque pueda haber una correlación entre número de genes y tamaño del genoma (es decir, que a mayor tamaño del genoma, mayor número de genes), esto no siempre es así. Por ello, a pesar de que tengamos organismos con un número elevado de genes, puede ocurrir que sus genomas no posean un gran tamaño, ya que poseen pocas regiones de DNA no codificante, y la mayoría de su genoma estaría formado por los genes que codifican proteínas (tendríamos de esta forma pocas regiones intergénicas entre cada uno de esos genes).
Número de genes y complejidad de los seres vivos
Aparte de la paradoja del valor C, cabe destacar que la idea de que el número de genes de una especie está relacionado con una mayor complejidad de la misma tampoco es cierta (11).
Esto es debido principalmente a que muchos genes se duplican, pudiendo sufrir una selección negativa hasta convertirse en los ya mencionados pseudogenes, o ir resistiendo a las duras exigencias de la selección natural y sufrir modificaciones (mutaciones) a lo largo del tiempo hasta dar lugar a un gen algo distinto al "original". De hecho, hay muchos genes redundantes que ejercen prácticamente la misma función, y por ello, si uno de esos genes redundantes sufriese una mutación que lo hiciese no funcional, no habría grandes alteraciones a nivel fenotípico (lo que se conoce como robustez), ya que si el otro gen sigue funcional, este puede realizar la misma función que ha dejado de desempeñar el otro.
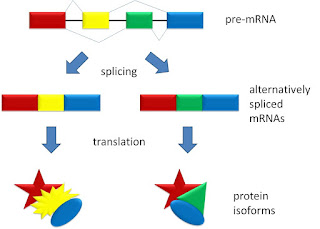
Además, existen mecanismos de economía genética como el splicing alternativo, que permite sintetizar más de una proteína a partir de un único gen a través de la modificación del pre-RNA mensajero (esto es lo que comenté al principio del post de que no es exactamente cierto que un gen codifica una única proteína). La economía genética es importante para la supervivencia de los organismos, ya que el hecho de poder sintetizar más de una proteína por gen evita que tengan que existir genes diferentes por separado para fabricar cada una de esas proteínas, lo cual supondría un mayor gasto energético y de material celular.
Esquema del splicing o ayuste alternativo: a partir de un transcrito de un gen, se pueden eliminar o conservar diferencialmente fragmentos concretos (exones) del pre-mRNA inicial, para dar lugar a distintos mRNA maduros, que obviamente codificarán polipéptidos diferentes (Fuente).
Esto hace que el número de genes no se corresponda exactamente con el número de proteínas expresadas en un organismo, las encargadas de realizar las funciones celulares, así que tampoco nos podemos guiar por el criterio del número de genes para describir la complejidad (término sujeto a interpretaciones) o sencillez de un organismo.
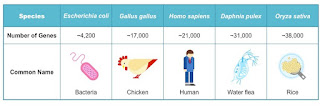
El número de genes de un organismo no es una buena forma de "medir" su complejidad. Habrá muchos/as que se estén llevando las manos a la cabeza al ver que el arroz que se comen en la paella tiene casi el doble de genes que ell@s.
Conclusiones
Además, la proporción variable de DNA no codificante en eucariotas hace que se produzca el fenómeno de la paradoja del valor C, por el que vemos que el nivel de complejidad de una especie no está correlacionado estrictamente con el tamaño de su genoma, el cual, a su vez, tampoco es un fiel reflejo del número de genes de un organismo (que tampoco es una buena forma de determinar si un ser vivo es más o menos complejo).
Así que, si alguien os acusa de ser personas demasiado "simples" o "complicadas", le mandáis este post y así le haréis ver que no es tan fácil determinar la complejidad de un ser vivo. ¡Que se habrá creído!
Isqui iris mi quimpliquidi, mimimimi...
Referencias
1. Epp, C. D. (1997). Definition
of a gene. In Nature (Vol. 389, Issue 6651, p. 537). https://doi.org/10.1038/39166
2. Portin, P., & Wilkins, A.
(2017). The evolving definition of the term “Gene.” Genetics, 205(4),
1353–1364. https://doi.org/10.1534/genetics.116.196956
5. Tutar, Y. (2012).
Pseudogenes. In Comparative and Functional Genomics (Vol. 2012). https://doi.org/10.1155/2012/424526
6. Rodic,
N. (2018). LINE-1 activity and regulation in cancer. Frontiers in Bioscience
- Landmark, 23(9), 1680–1686. https://doi.org/10.2741/4666
7. Kerachian,
M. A., & Kerachian, M. (2019). Long interspersed nucleotide element-1
(LINE-1) methylation in colorectal cancer. In Clinica Chimica Acta (Vol.
488, pp. 209–214). https://doi.org/10.1016/j.cca.2018.11.018
9. Gil,
R., & Latorre, A. (2012). Factors behind junk DNA in bacteria. In Genes
(Vol. 3, Issue 4, pp. 634–650). https://doi.org/10.3390/genes3040634
10. Eddy,
S. R. (2012). The C-value paradox, junk DNA and ENCODE. In Current Biology (Vol. 22, Issue 21). https://doi.org/10.1016/j.cub.2012.10.002
11. Gerbi,
S. (2012). Eukaryotic Genome. Nature Education, 2012(I), 2050.






No hay comentarios:
Publicar un comentario